研究者の荒川央さんと新田剛さんの間でDNAプラスミド混入を巡って論争が起きています。今回荒川さんがNOTEに出した論考から、何処に違いがあるのか、何を巡って相違があるのかを明らかにしています。当方は技術的な中身についてさっぱり分りませんが、二人の違いについて分りやすいのではないかと思い、紹介します。

定量PCR再々考 (新田剛先生に向けて)
DNA混入についての記事を掲載して以来、私のブログもかつてなかったほどの攻撃を受けています。今回の記事は大変長いものになりますが、自衛の意味も含め、今一度私がDNA混入問題について考えている事を忌憚なく述べさせていただこうと思います。
改めて経緯を説明させていただきます。事の発端は、「RNAコロナワクチンにDNAが混入している?」の記事を書いた約1週間後の2023年4月11日に、新田剛先生から突然メールが送られて来た事です。それまで私は新田先生とは勤務医団や副反応検討会のオンラインミーティング等で一言二言言葉を交わす機会があった程度で、特に交流はありませんでした。
メールはコロナワクチンのDNA混入についてでした。内容は、「私 (新田) は既に自分自身で実験をしてデータも持っている。あなた (荒川) はMcKernan氏のデータを検証しているか? もしくは他の研究者からの追試の情報を持っているか?」でした。また、「McKernan氏の発信だけを根拠にしているなら、この話の信頼性が揺らぎ、足を掬われることになりかねない」といった言葉もありました。正直私にはこの「足を掬われる」の意味が分かりませんでした。一体誰が足を掬いに来るのか。
私自身としてはもちろんコロナワクチンの内容物については以前から大きな関心がありましたが、コロナワクチンの成分分析は立場によってはそれ自体が法に触れる可能性もあると理解しており、実際に現物を調べた事などありませんでした。そして入手自体もまず不可能です。ですから、そのような内容のメールが送られて来た事に大変困惑しました。
いずれにせよ、そもそもほとんど知らないような方と秘密裏に法律に抵触する恐れもあるような研究のやり取りなど私にはできません。自分自身もその行為に加担する事になるからです。また、他の研究者の実験内容についても、仮に私が知っていたとしても本人の了承無しに話したりなどできるわけがありません。ですから、「新田先生が本当にこの件の話をしたいのであれば、オープンの場でやりとりしましょう。」という意思表示も込め「ブログにコメントして聞いてください」と返信しました。
実際、私の元にはブログの内容やコロナワクチンに関する科学的な質問等の問い合わせが多く来るのですが、その都度クローズドで個々のやりとりを繰り返すのは時間も労力も削られてしまいます。そのため、より多くの人たちと情報を共有した方が建設的であるという考えの元、基本的にはそういった問い合わせには「ブログのコメント欄に質問として書き込んでください」と返すようにしているという事情もあります。
以下は新田先生への私からの返信です。
新田先生
ご心配を賜りありがとうございます。
私は基本的にコロナ関係のメールでの質問にはメールでそのまま返答するのではなく、改めてnoteブログのコメント欄に内容を書き込んでもらう方針を取っております。ブログ内容等について日々多くのメールが来ており、その都度クローズドの場での対応をするより、重要な情報を大勢の人と共有するためです。
よろしければ、同じ質問を私のnoteブログのコメント欄に書き込んでいただけるでしょうか。
https://note.com/hiroshi_arakawa/n/n2b4f8555b103実際に多くの方の興味の対象でしょうし、公開で討論した方が良いですし、建設的だと思うのです。専門的な質問ですので、その際には所属、氏名を明記されるように重ねてお願い致します。
よろしくお願い致します。
荒川 央
それを受けた新田先生は、以下のようにコメント欄に書き込まれました。
荒川先生
東京大学の新田 剛です。
私の知る限りでは、mRNA-LNP製剤にDNA断片が混入しているのは間違いありませんが(というか周知の事実です)、プラスミドDNAの混入は検出されていません。Kevin博士も、カナマイシン寒天培地上の大腸菌コロニーからプラスミドDNAを回収できていません。したがって「プラスミドDNAの混入」は証明されていません。
荒川先生は、ご自身でKevin博士の実験データを追試しておられますでしょうか? あるいは、他の研究者からの追試実験の情報をお持ちでしょうか?
もしKevin博士が発信していることだけを根拠にされているようでしたら、この話の信頼性が揺らぎ、足を掬われることになりかねないと危惧しております。もし誰かが、mRNA-LNP製剤中に完全長のプラスミドDNAがある、という証拠を提示しているのであれば、状況は大きく変わります。そのプラスミドDNAを、ヒト細胞やマウスに打ち込んで何が起きるか、を検証する実験に進めるからです。しかし現状では、「プラスミドDNAの混入」の最初の段階でつまずいていると思います。
さて、新田先生からのメールには彼の実験結果も添付されており、そこには彼自身の解釈も記されていましたが、その実験結果についての私自身の解釈は全く別のものでした。そして、この解釈次第では彼の実験結果自体がコロナワクチンへのDNA混入を裏付けているとも受け取れるのです。それについてのお話しをしていきたいと思います。
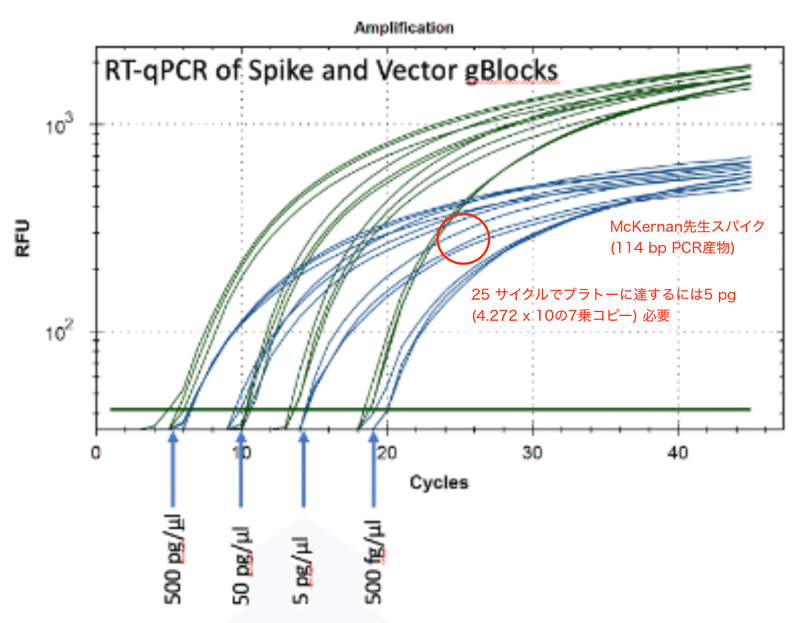
図1はMcKernan先生のqPCRです。Ct値とはcycle threshold value、qPCRで設定した閾値に達するqPCRのサイクル数です。この閾値は機械で自動的に設定される場合もあれば、手動で任意に設定する事もできます。極端な話、DNA (実際には検出される蛍光シグナル) が指数的に増幅している領域のどこに設定しても対照実験とサンプルの比較は可能です。
対数目盛を使うか、線形目盛 (対数でない普通の目盛) を使うかは機器にもよりますし、どちらを使うかは目的次第であり、ある意味「好みの問題」です。対数は数桁異なるような非常に大きな差を比べたり、指数的に増幅するものをグラフに表示するのに便利です。対数には0がありませんが、線形グラフなら0を表示できます。qPCRに限らず、私自身もそれぞれの実験の目的次第で対数グラフ、線形グラフを使い分けており、どちらを使わなければいけないというものでもありません。
同一の実験内でのCt値の比較は難しくありません。しかし、別の機器による別の実験を比べるのは容易ではないのです。qPCRの機器や解析ソフトウェアが異なればPCRの立ち上がりやCt値も異なります。機器やソフトウェアによって閾値が変わる事もありますし、また、閾値を任意に変更する事も可能です。しかし、プラトーの位置は基本的にそうした要素の影響を受けません。そのため、プラトーに達するのに必要なサイクル数を比べる事で別々の実験のPCRの効率を比較する事が可能となります。
濃度既知のレファレンスを見てください。図1のグラフの下のDNAの量の表記には「/µl」の表示がありますが、McKernan先生にその意味を問い合わせてみたところ、それぞれの濃度のDNAを1 µl使用したとの事でした。DNAの大きさ (長さ) の単位はbp (base pair = 塩基対) です。DNA断片長に応じて質量当たりのコピー数は変わりますが、以下のwebサイトなどで計算する事ができます。
dsDNA: Mass to/from Moles Convertor
https://nebiocalculator.neb.com/#!/dsdnaamt
図1ではPCR産物が最大量近くに達してもPCR産物は徐々に増え続けています。qPCRは感度が良いため、プライマーやPCR産物の配列次第ではこういう事もあります。
25サイクル辺りに注目してください。25サイクルでプラトーに達しているのは出発材料5 pg (4.272 x 10の7乗) のDNAです。
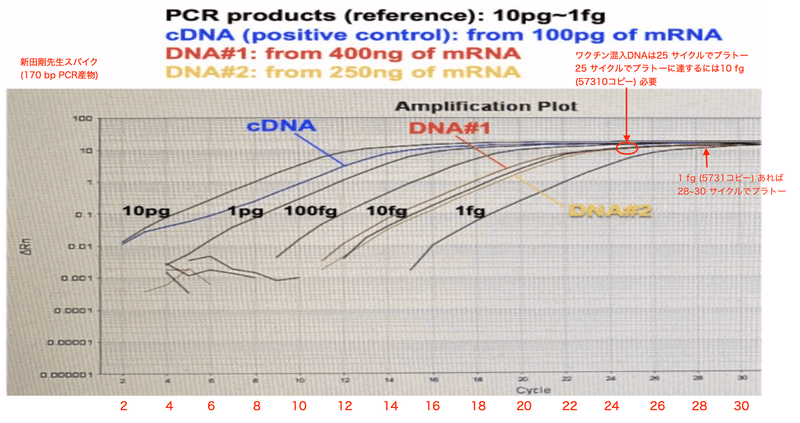
図2はワクチン混入DNAについての新田先生自身のqPCRの結果です。これは新田先生ご本人がSNS上でも公開されたデータですので、既に公共のデータとしての扱いをして良いものであると考えます。
図を見るとワクチン混入DNAは25サイクル程でプラトーに達しています。問題は、混入DNAと同じく25サイクルでプラトーに達しているレファレンスが10 fgだという事です。1 fgのDNAも25サイクルの時点で大きく増幅がかかっており28サイクルではほぼプラトーに達しています。
プラトー到達25で比較すると、新田先生の実験では 10 fg (57310コピー)、McKernan先生の実験では 5 pg (4.272 x 10の7乗) と1000倍程レファレンスの増幅が異なります。ここで一番の疑問は、「なぜ濃度既知のレファレンスの増幅の速さがこれほど異なるのか?」という事です。qPCRは感度の高い実験ですが、PCRの条件が異なるだけでそれほど差が出るものでしょうか?
その点が気になったため、武漢型コロナのスパイク遺伝子を含むプラスミドを使って私自身でも検証の為の実験を行いました。この実験はコロナワクチンに混入したDNAの検出実験の追試ではなく、その対照実験となるレファレンスについての追試です。PCRの鋳型には、新田先生、McKernan先生のそれぞれのPCRアンプリコンと同じサイズ、ほぼ同じ配列のDNAを用いました。用いたプライマーも新田先生、McKernan先生が用いたものと同じものです。
私が用いたプラスミドはpCEP4-myc-S (Wuhan variant) です (武漢型スパイク遺伝子を含む)。このプラスミドの武漢型スパイク遺伝子はコドン最適化によりオリジナルのスパイク遺伝子から配列が変更されているのですが、ファイザー、モデルナのスパイク遺伝子のどちらとも少しずつ配列が異なり、プライマーが対応する位置にも配列の違いが少しありました。そのため、一度PCRで増幅しました。そうするとプライマーの位置の変異はプライマーと置きかわります。以降、このPCR産物を鋳型として用いました。
McKernan先生はPCRのプライマー配列を公開しています。
Spike_Forward: AGATGGCCTACCGGTTCA
Spike_Reverse: TCAGGCTGTCCTGGATCTT
新田先生が実験で使われたプライマー配列は私が知る限りオープンの場では公開されておりませんが、メールには書かれていましたので、私は同一の配列のプライマーを用いて実験しました。また新田先生は、「McKernan先生のプライマーは短い」とも批判していましたが、新田先生自身のプライマーもMcKernan先生のものとほぼ同じ長さのものでしたので、新田先生の計算間違いでないとすれば、その批判自体が自己矛盾しています。ちなみに新田先生のプライマーはどちらも19塩基でしたが、McKernan先生のプライマーは18塩基と19塩基です。1つのプライマーが1塩基短いだけです。
使用したのはLuna Universal qPCR Master Mix (NEB, M3003S)。機器はRocheのLightCycler 96です。プロトコルは「95°C, 1 min → (95°C, 15 s → 60°C, 30 s) x Xサイクル」。キットで推奨されているプロトコルを使用しました。機器はRocheのLightCycler 96です。
以降の実験データはMcKernan先生、新田先生のレファレンスのPCRについての追試であり、私自身で行った実験の結果です。
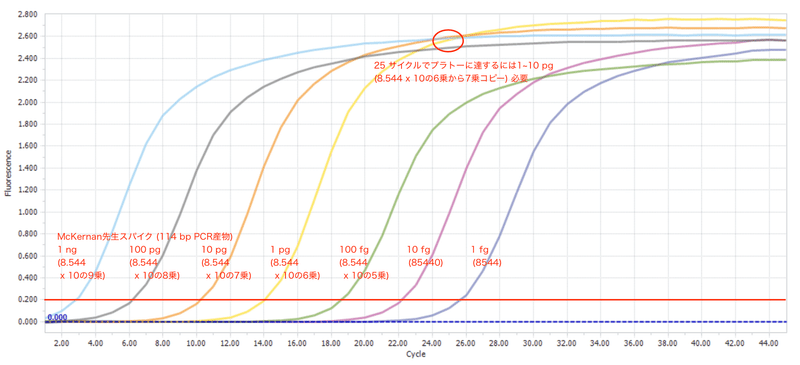
図3はMcKernan先生のレファレンスの追試実験を私自身で行ったものです。25サイクルでプラトーの頂点近くに達するには出発材料が1〜10 pg必要でした。これは8.544 x 10の6乗から7乗コピーに当たります。
この実験で私が使ったのはRocheのLightCycler 96です。増幅曲線の結果は線形目盛で表示されます。
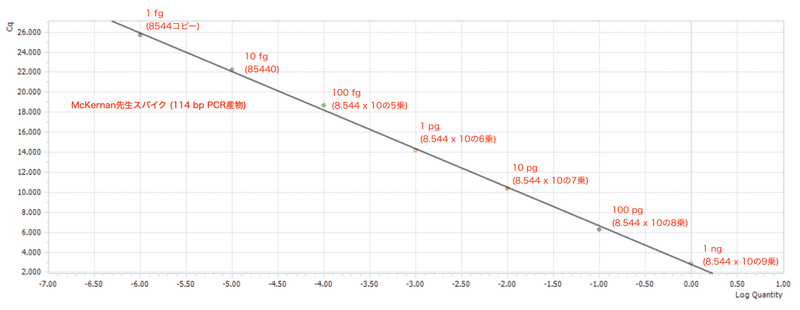
図4は図3の検量線です。
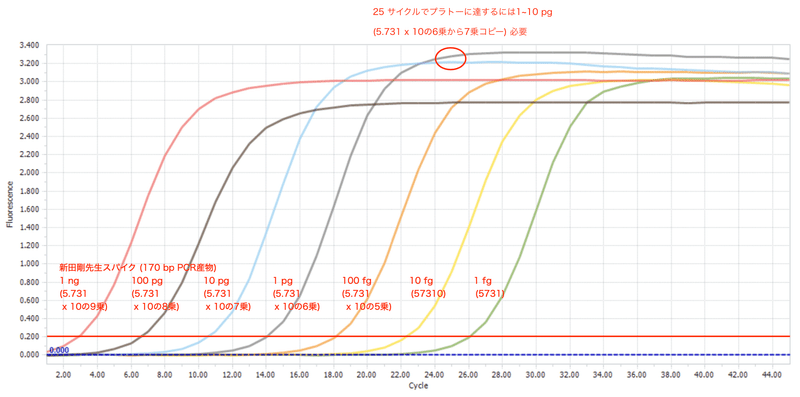
図5は新田先生のレファレンスの追試実験を私自身で行ったものです。25サイクルでプラトーの頂点近くに達するには出発材料が1〜10 pg (5.731 x 10の6乗から7乗コピー) 必要でした。
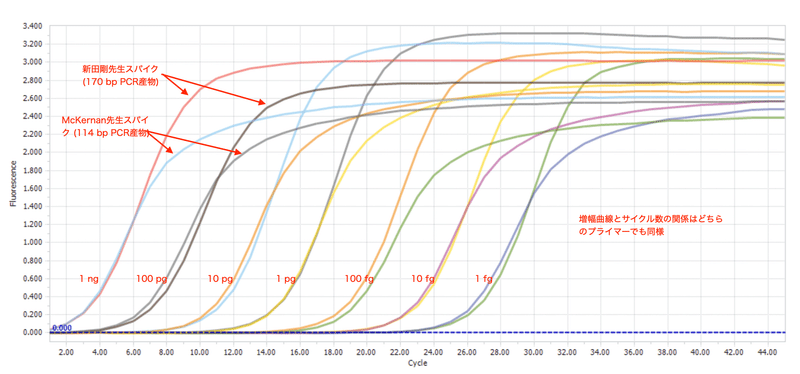
図6はMcKernan先生、新田先生のレファレンスの追試実験を重ね合わせたものです。PCRの効率はほぼ同じでした。プラトーの高さが若干異なるのはPCR産物の大きさの違いのためでしょう。
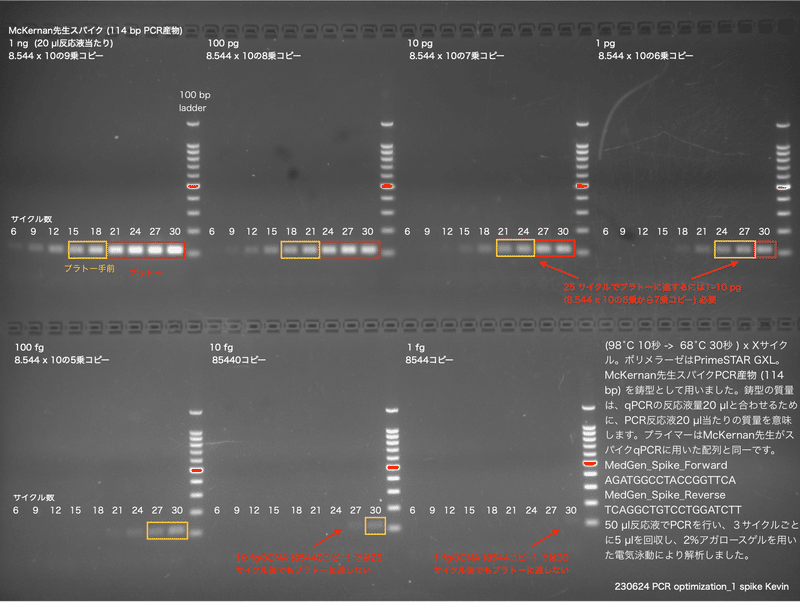
図7は私が同様の実験をqPCRではなく通常のPCRで再現し、アガロースゲルによる電気泳動で解析したものです。PCRを用いた理由はプラトーに至るサイクル数を別の実験で再確認するためです。電気泳動の解析ではDNAを大きさによって振り分けるので、正しいPCR産物とアーティファクト (間違い産物) を大きさで区別できます。臭化エチジウムを用いた染色は、蛍光を用いるqPCRほど感度が良くないため、PCRの立ち上がりは遅くなりますが、むしろ感度が比較的低いために、プラトー辺りを確認するにはqPCRよりも通常のPCRの方が視覚化しやすい利点があります。
私は自分自身の研究で、しばしばライブラリDNA (多様なDNAの集まり) のPCR増幅を行います。プラトー以降はライブラリのDNA構造が変化するのですが、アガロースゲル電気泳動ではその確認が容易なため、プラトー辺りを観察するために図7のような半定量PCR実験を行う事が多いのです。
増幅用に使った酵素はPrimeSTAR GXLです。(98˚C 10秒 → 68˚C 30秒) のサイクルを30サイクルまで繰り返しました。鋳型の質量は、qPCRの反応液量20 µlと合わせるためにPCR反応液20 µl当たりの質量を意味します。50 µl反応液でPCRを行い、6サイクルから始めて、3サイクルごとに5 µlを回収し、2%アガロースゲルを用いた電気泳動により解析しました。
PCR産物が最大量近くまで増えた状態を赤で、その少し前の状態を黄色で囲みました。25サイクルでプラトーに達するには1〜10 pgのDNAが必要でした。コピー数では10の7乗から8乗ほどです。ちなみに1 fgのDNA (8544コピー) では30サイクルの増幅でもプラトーに届きません。
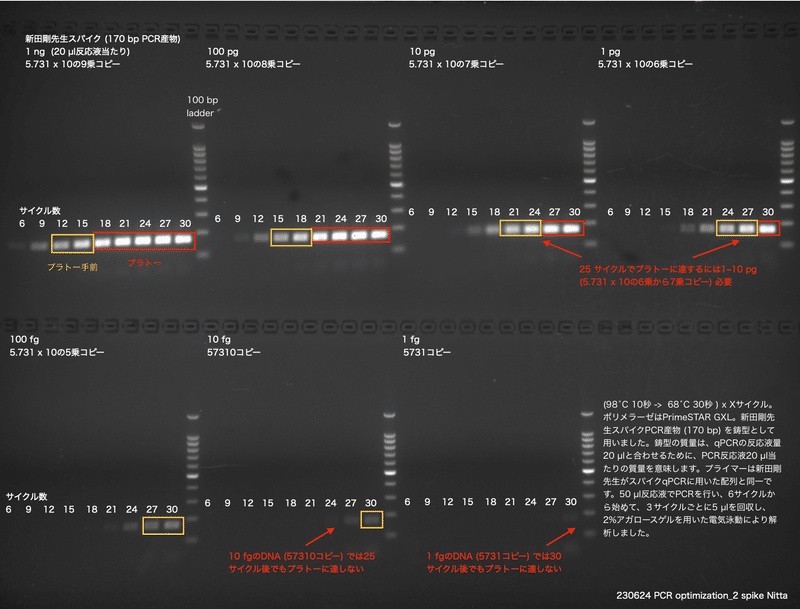
図8は新田先生が使ったのと同じPCRプライマーによるスパイク遺伝子のPCRを私が半定量PCRで再現したものです。プライマー以外のPCRの条件は図7と同一です。新田先生のPCR産物がより明るいのは、DNAの大きさが大きいために、DNA一分子当たりに結合し、光る素材 (臭化エチジウム) の量が多くなるためです。
25サイクルでプラトーに達するにはやはり1〜10 pgのDNAが必要です。コピー数では5 x 10の6乗から5 x 10の7乗ほどです。ちなみに1 fgのDNA (5731コピー) では30サイクルの増幅でもプラトーに届きません。
私の今回の実験や今までの経験からも、10000コピー以下のDNAでは30サイクルではプラトーには到底届きません。今回の実験でも25サイクルでプラトーに達するには新田先生のプライマーでもMcKernan先生のプライマーでも1〜10 pg (5 x 10の6乗から10の8乗コピー) のDNAが必要でした。これに対し、図2の新田先生のqPCRでは10 fg (57310コピー) で十分でした。
私の結論は、「新田剛先生はレファレンスのDNA量を大幅に過小評価している可能性が極めて高い」という事です。具体的な過小評価の程度は100〜1000倍です。つまりそれは、コロナワクチンに混入しているDNAの量が新田先生の主張するよりもそれだけ多い可能性を意味します。

表1はMcKernan先生と新田先生の実験手法及び行動とその影響をまとめた対照表です。
McKernan先生はゲノム解析の専門家です。ディープシークエンシングのコストは年々下がってはいるものの、基本的に高価な実験であり、1回の実験当たり数十万〜百万円かかる事も多いです。出発材料のDNAの品質が悪ければ実験は台無しになるので、高品質のDNAを調整する必要があります。そのためゲノム解析の専門家は高品質のDNAを調整する経験や知識が豊富です。実際、McKernan先生は混入DNAのロスを最小限にする工夫をし、細心の注意を払ってLNPなどの定量法を阻害する物質を取り除いています。混入DNAの配列を明らかにするために、ディープシークエンシングによって網羅的塩基配列解析をしており、混入DNAの断片長はAgilentテープステーションによって解析しています。シュードウリジンがどのように定量の障害になるのかも分かりませんので、McKernan先生はQubit蛍光光度計、Agilentテープステーション、qPCRの3種類の異なる定量法を用いて混入DNAの定量を行いました。
ちなみに、McKernan先生が混入DNAの定量データを取り下げたとして、悪意ある曲解を流している人も見受けられますが、それは正しくありません。ブログ記事もデータも今現在もそのまま記載されています。そして、一般論として同一プレプリント内でデータを取り下げたりはしません。
qPCRはDNAを増幅し、その増幅具合によって定量するという間接的定量法です。増幅阻害物質の影響を受けますし、損傷を受けたDNAの定量には不向きです。McKernan先生はqPCRを定量化よりもむしろ、なぜコロナワクチンにDNAが混入してしまうかの機序の解明に応用し、その理由まで明らかにしました。シュードウリジン化RNAがDNAに強固に結合し、DNase Iによる分解からDNAを保護していたのです。これはmRNAワクチン製造の際の致命的な欠陥とも言えます。
McKernan先生とも何度かメールでやりとりしましたが、彼にとってもLNPやシュードウリジン化RNAは「未知の素材」です。McKernan先生の専門はゲノム解析であり、mRNAワクチンではありません。McKernan先生はコロナワクチン混入DNAの精製法、定量法、解析法、ディープシークエンスデータまで公開しています。そしてどれも緻密に構成された実験デザインです。McKernan先生の実験は精製、解析のレベルが高く、また混入DNAを検出するために最善の努力を払っています。
McKernan先生はコロナワクチンに混入したDNAが大腸菌を形質転換できる事を発見しました。そしてこれが環境由来の抗生物質耐性菌が偶然混入したものではない事も実験で確認しています。コロナワクチンに混入したDNAが大腸菌に導入されて、カナマイシン (抗生物質) 耐性になっています。ただ、環状の全長プラスミドの存在については結論が出ていません。新田先生は全長環状プラスミドDNAの検出について繰り返し言及していましたが、それは本質的な問題ではありません。「プラスミドの全長配列を再現できるだけのDNA断片がワクチンから検出された事」自体が大変な驚きであり、深刻な問題なのです。そもそもLNPに包まれたRNAを大規模に人間に投与した事など史上初の試みであり、しかも、その中にDNAが混入していたのです。長期の安全性のデータ自体が存在しないために、人体にとって安全な量の基準など実際にはまだ分からないのです。
大腸菌で増殖できるプラスミドDNAは環状のものです。ワクチン混入DNA内に環状プラスミドが存在しない場合、事態はさらに深刻な可能性があります。大腸菌がカナマイシン耐性になっているのなら、カナマイシン耐性遺伝子が環状プラスミド以外のどこかにあるはずです。菌内で遺伝子が安定に存在できる場所はプラスミド以外では大腸菌のゲノム内です。つまり、カナマイシン耐性遺伝子が大腸菌ゲノムに組み込まれた可能性があるという事です。形質転換の実験では時折こういう事が起こります。全長の環状プラスミドでなくとも、プラスミドの一部が大腸菌ゲノム内に取り込まれ、さらに遺伝子として機能したならば、それはまさにmRNAワクチンの潜在的リスクの根拠となります。なぜならLNPによってDNAはヒト細胞内に取り込まれますし、SV40プロモータによって核に輸送され、ヒトゲノムに取り込まれる過程と同様の事態だからです。
繰り返しますが、McKernan先生は実験手法の詳細を公開しています。にも関わらず新田先生はあえてそれとは異なった手法で実験をしました。新田先生はMcKernan先生とは異なるプライマーを用いています。そしてアンプリコンが大きくなるために、損傷を受けたDNAからは増幅がかかりにくくなる実験デザインを組んでいます。また、プライマーの配列を含めて実験の基本条件を公開していません。精製法においてDNAのロスを最小限にする工夫が足りず、定量法を阻害する物質を除く工夫も見られません。実際、LNPやシュードウリジンがどのように精製や定量の障害になるのかは分かりません。高品質のDNAが精製できるとも考えにくく、どれくらいのDNAがロスされるかも分からない。端的に言って、新田先生の混入DNAの精製法は非常に「雑」なのです。圧倒的に解析が足りません。むしろ混入DNA量を「過小評価するための最善を尽くしている」ようにすら見えるのです。同様の感想をMcKernan先生も持っていました。新田先生の定量はqPCRでしか行われていませんが、qPCRはそもそもワクチン混入DNAの定量には至適な方法ではありません。しかもレファレンスDNAの増幅度が奇妙で、信頼できるデータとは考えにくいのです。
さらに言うならば、McKernan先生と新田先生の実験の根本的な違いは技術レベルです。McKernan先生の実験は最新のゲノム解析であり、実験方法も緻密に構成されています。これに対し新田先生の実験は、古典的かつ初歩的な技術レベルのものであり、実際のところ大学理学部の4年生が実習で行うようなごく基礎的な実験です。このような内容の実験では、コロナワクチンへの混入DNAの量が少ない証明とはなり得ません。不十分なデータをもって他者のデータを否定する事はできないのです。他者の研究を「否定」するには、質的にも量的にも確かな根拠が必要となります。このような実験を根拠に、「混入DNAはわずかなので騒ぐべきではない」などとは到底言えない、という事です。
また、コロナワクチンはロットごとの差が大きいですので、混入DNA量もロットごと、バイアルごとに大幅に異なっている可能性を考慮すべきでしょう。仮に数個のバイアルを調べ、その際の混入DNA量が少なかったとしても、他のバイアルや他のロットの混入DNA量も同様に少ないなどとは決して断定できません。
そして、私が今回新田先生の行動で特に大きな問題と感じているのは、McKernan先生の結果を「STAP細胞的な匂いを感じる」「与太話」と蔑んだり、「それに乗って情報発信されるのは危険」などと他者を牽制しようとしている事です。

本当の「スキャンダル」なら、複数の研究者が複数の方法で検証し、そのデータをもとに全ロットの分析や回収に向けて研究機関や行政に働きかけるべきです。
もし1人の研究者が書いたブログ記事だけが根拠なら、「与太話」である可能性すらあります。それに乗って情報発信されるのは危険だと申し上げております。
https://note.com/hiroshi_arakawa/n/nfde06a8f138b
McKernan先生は大学院を中退していますが、在学中に自身の発明した技術を元に起業し、会社を大きく育てられました。これは並大抵な事ではありません。ディープシークエンサーの開発者であり、複数の特許を持ち、自らゲノム解析の専門家です。つまりある意味、利益相反とは真逆の立場です。コロナワクチンへのDNAの混入は紛れもなく大きなスキャンダルです。場合によっては自身が「反ワクチン」のレッテルを貼られ、評判を落として自社の売り上げを落とす恐れさえあるのです。また、もし仮にゲノム解析の会社がこうした件での「捏造」が発覚でもしたならば、会社自体が倒産する恐れすらあります。そのような立場であるにも関わらず、専門家中の専門家が危機を真剣に訴えている。McKernan先生は実際それだけのリスクを背負って行動されており、この背景の意味は大きいと私は考えます。
McKernan先生はコロナワクチンへのDNA混入の追試を世界に呼びかけています。現在までのところ、オランダのグループもDNA混入検出に成功しています。実は彼らは2021年の時点ですでにその結果を得ていました。私も彼らと連絡を取り確認したのですが、彼らはディープシークエンシング解析まで行っています。しかしながら、コロナワクチンのワクチン接種以外の使用目的が規制されているためにデータを公表する事ができないのです。また、米国の別のグループもMcKernan先生が発見した混入プラスミドのDNA配列を確認しました。今後も世界中で検証は続くでしょう。直近ではMcKernan先生はFDAの委員会でもコロナワクチンへのDNA混入を告発し、またオーストラリアの法廷でもDNA混入についての証言を求められています。McKernan先生はコロナワクチンへのDNA混入を確信し、勢力的に活動され続けています。
さて、私自身はかねてより、新田先生といつかの時点ではオープンの場で直接お話すべきであろうと考えておりました。そして、その気持ちの中でこの記事を書いていました。
そんな折、Trilliana華さんがTwitterのスペースでの新田先生との対談相手として私に打診をされました。
これも良い機会だと私は考えました。
改めまして、新田剛先生、もし反論がおありでしたら直接対談で討論しませんか?
新田先生もお忙しい事と存じますし、私自身も多忙です。直接の対談なら1、2時間で済むでしょう。対談が実現する事になれば、技術的な点に加え、お聞きしたい事がたくさんあります。また、この件に関しましては、今後も経緯を含め全てオープンでのやりとりとさせていただきたく存じます。よろしくお願い致します。
